Contexte :
La transplantation hépatique est une solution de dernier recours pour les patients en insuffisance hépatique terminale. Cependant, en raison de la pénurie de greffons, les donneurs à critères élargis (âgés, stéatosiques, ou ayant subi une ischémie) sont de plus en plus sollicités. Ces greffons, bien qu'indispensables pour réduire les listes d'attente, présentent des risques plus élevés de complications post-transplantation, en particulier lorsqu'ils sont affectés par la stéatose hépatique. Celle-ci se manifeste par une accumulation de graisses dans les hépatocytes, se divise en micro-stéatose (gouttelettes de petite diamètre) et macro-stéatose (gouttelettes plus grandes), cette dernière étant particulièrement critique pour la viabilité du greffon.
Aujourd'hui, l'évaluation de la stéatose repose principalement sur des biopsies et des analyses histopathologiques, une méthode invasive, subjective [3] et ne fournissant qu’une évaluation locale du foie. Afin d’apporter une solution plus rapide et non invasive, des mesures de réflectance diffuse en infrarouge ont été récemment effectuées sur des échantillons de foie dans le cadre de la thèse d’Antoine Uzel [1], [2]. Cette méthode optique permet d’obtenir des informations sur les propriétés des tissus sans recours à la biopsie, offrant ainsi une alternative prometteuse [4].
Objectifs du stage :
Ce stage vise à développer une méthode non invasive pour évaluer la micro et macro-stéatose hépatique via des simulations Monte Carlo et un algorithme d'apprentissage profond. Le/la stagiaire participera à toutes les étapes du projet, de la modélisation théorique à la validation expérimentale.
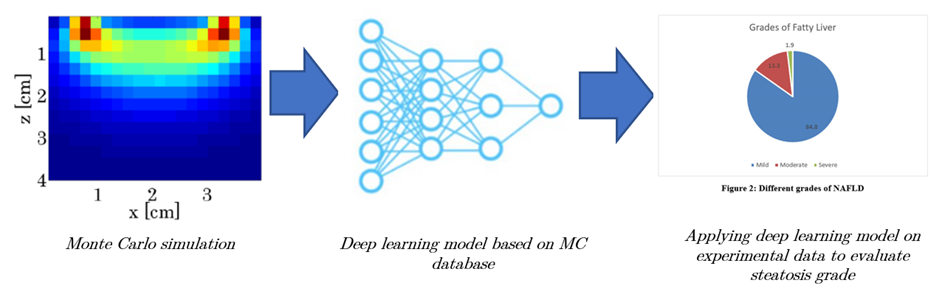
1. Simulations Monte Carlo :
Utiliser les simulations Monte Carlo [5] pour modéliser la réflectance diffuse dans le foie avec différentes conditions de micro et macro-stéatose. Ces simulations fourniront une base de données de spectres infrarouges dépendant du taux de stéatose du foie.
Objectifs clés :
- Modéliser la réflectance diffuse sous différentes conditions de stéatose.
- Étudier l'impact des variations de taille et de densité des gouttelettes lipidiques.
2. Entraînement d’un modèle de deep learning :
Entraîner, de manière supervisée à partir des spectres générés par les simulations, un modèle d'apprentissage profond de type Graph Neural Network [6], [7] pour prédire les taux de stéatose. L'algorithme devra être capable de distinguer la micro et la macro-stéatose en analysant les spectres infrarouges.
Objectifs clés :
- Entraîner un modèle à partir des données simulées.
- Optimiser le modèle pour améliorer la précision des prédictions de stéatose.
3. Validation expérimentale :
Tester le modèle sur des spectres réels acquis sur des échantillons biologiques (souris ou fantômes) et comparer les résultats aux analyses histopathologiques afin de valider la fiabilité de la méthode.
Objectifs clés :
- Comparer les résultats de l’algorithme avec les données de référence histopathologique.
- Possibilité d’acquisition de données in-vivo (souris/greffon humain) ou sur fantômes si souhaité par le candidat.
Profil recherché :
Le/la candidat(e) devra avoir de bonnes compétences en modélisation numérique et en traitement de données, ainsi qu’une connaissance des algorithmes d’apprentissage automatique. Une expérience préalable en simulations Monte Carlo, analyse de spectres ou optique biomédicale serait un plus.
- Connaissance de programmation sur Python (connaissance de Pytorch, TensorFlow serait un plus)
- Connaissance de Matlab serait un plus
- Intérêt pour l’optique biomédicale et l'apprentissage profond.
Compétences développées pendant le stage :
- Maîtrise des techniques de simulation Monte Carlo pour la modélisation des interactions lumière-tissu.
- Développement de compétences avancées en apprentissage profond et traitement de données biomédicales.
- Analyse et traitement de données expérimentales issues de spectrométrie infrarouge.
Informations complémentaires :
- Durée : 4 à 6 mois
- Localisation : Laboratoire CREATIS, 21 Avenue Jean Capelle O, Villeurbanne
- Encadrement : Cédric Ray-Garreau (CREATIS), Bruno Montcel (CREATIS), Antoine Uzel (CREATIS)
- Candidature : Envoyer CV, relevé de notes et lettre de motivation à cedric.ray[at]univ-lyon1.fr, bruno.montcel[at]univ-lyon1.fr et antoine.uzel[at]creatis.insa-lyon.fr
[1] A. Uzel, M. Sdika, S. Chopinet, O. Lopez, et B. Montcel, « Near infrared diffuse reflectance spectroscopy for fat quantification in non-alcoholic fatty liver disease », in Diffuse Optical Spectroscopy and Imaging IX, SPIE, août 2023, p. 117‑120. doi: 10.1117/12.2669268.
[2] A. Uzel, M. Sdika, S. Chopinet, O. Lopez, et B. Montcel, « Near infrared diffuse reflectance spectroscopy for fat quantification in fatty liver disease », in Advanced Biomedical and Clinical Diagnostic and Surgical Guidance Systems XXII, SPIE, mars 2024, p. 54‑61. doi: 10.1117/12.3001895.
[3] D. A. H. Neil, M. Minervini, M. L. Smith, S. G. Hubscher, E. M. Brunt, et A. J. Demetris, « Banff consensus recommendations for steatosis assessment in donor livers », p. 12.
[4] R. Nachabé, Diagnosis with near infrared spectroscopy during minimally invasive procedures. 2012. Consulté le: 6 septembre 2023. [En ligne]. Disponible sur: https://repub.eur.nl/pub/32630/
[5] S. A. Prahl, « A Monte Carlo model of light propagation in tissue », présenté à Institutes for Advanced Optical Technologies, G. J. Mueller, D. H. Sliney, et R. F. Potter, Éd., Berlin, Germany, janv. 1989, p. 1030509. doi: 10.1117/12.2283590.
[6] Wu, Z., Pan, S., Chen, F., Long, G., Zhang, C., Yu, P.S., 2021. A Comprehensive Survey on Graph Neural Networks. IEEE Transactions on Neural Networks and Learning Systems 32, 4–24. https://doi.org/10/ggrj8p
[7] Zhou, J., Cui, G., Hu, S., Zhang, Z., Yang, C., Liu, Z., Wang, L., Li, C., Sun, M., 2020. Graph neural networks: A review of methods and applications. AI Open 1, 57–81. https://doi.org/10/gjt96x
