Contexte :
Les méningiomes sont les tumeurs cérébrales primitives les plus courantes, et leur traitement nécessite une délimitation précise des composants tumoraux sur les images d'IRM. La segmentation automatisée des méningiomes à l'aide de techniques d'apprentissage profond représente un défi passionnant, étant donné la diversité morphologique et l'emplacement varié de ces tumeurs dans le cerveau. Ce stage vise à tester et adapter des méthodes d'intelligence artificielle développées pour les gliomes, afin d'améliorer la segmentation des méningiomes.
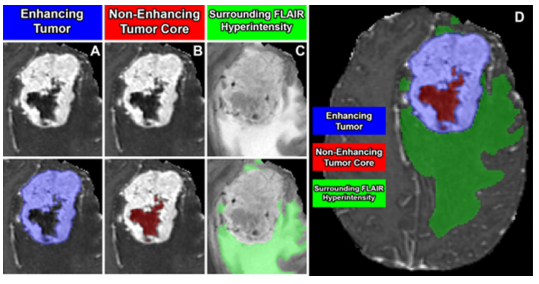
Fig : Les sous-compartiments des méningiomes. Les panneaux d'images A-C indiquent les différents sous-compartiments tumoraux ; (A) tumeur rehaussée (bleu) visible sur une image T1 pondérée après contraste ; (B) le noyau tumoral non rehaussé (rouge) visible sur une image T1 pondérée après contraste ; (C) l'hyperintensité FLAIR environnante (vert) visible sur une image T2/FLAIR pondérée ; (D) segmentations manuelles combinées générant les étiquettes finales des sous-compartiments tumoraux.
Objectifs du stage :
- Étude de l'État de l'Art : Se familiariser avec les techniques de segmentation existantes pour les gliomes et les méningiomes, en mettant l'accent sur les méthodes utilisant les réseaux de neurones convolutifs (CNN, Unet, nn-Unet).
- Adaptation de modèles : Développer des compétences en transfert de domaine en adaptant un modèle pré-entraîné pour les gliomes, riche en données annotées, pour qu'il puisse fonctionner efficacement sur les méningiomes, où les données annotées sont rares et présentent des différences (localisation extra-axiale, morphologies variées). Etude de méthodes telles que le fine-tuning pour réentraîner partiellement le modèle avec les données du domaine cible et exploration d'approches visant à aligner les distributions des deux domaines.
- Évaluation de Performances : Analyser les performances des modèles sur un ensemble de données d'IRM multi-séquences de méningiomes, en évaluant des métriques clés telles que le coefficient de Dice et la distance de Hausdorff pour quantifier l'exactitude de la segmentation.
Méthodologie :
- Acquisition de Données : Utilisation de jeux de données provenant du défi BraTS 2023 sur les méningiomes, contenant des IRM multi-séquences (T1, T2, FLAIR, T1Gd).
- Environnement de Développement : Mise en place d'un environnement de développement basé sur Python, avec des bibliothèques de deep learning telles que TensorFlow ou PyTorch.
- Collaboration : Travailler en étroite collaboration avec une équipe multidisciplinaire composée de chercheurs en imagerie médicale, experts en IA, et cliniciens.
Profil Recherché :
Étudiant en imagerie médicale, informatique, ou domaine connexe, avec un intérêt marqué pour l'apprentissage automatique. Des compétences en programmation (Python) et une compréhension des concepts de base en imagerie médicale seront un atout.
Bénéfices :
Ce stage offre l'opportunité de contribuer à des recherches à la pointe de la technologie en imagerie médicale, d'acquérir des compétences pratiques en intelligence artificielle, et d'explorer un domaine à fort impact sur le traitement et la gestion des tumeurs cérébrales.
